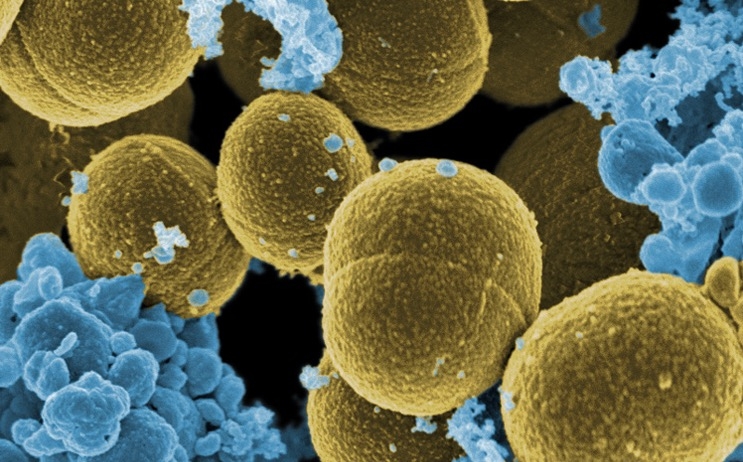
El 10% de las muertes en hospitales europeos por bacterias resistentes ocurre en España. Tenemos un problema porque consumimos más antibióticos de los necesarios. Los patógenos se acostumbran, mutan y sólo quedan vivos los más fuertes, que siguen reproduciéndose. Afortunadamente, conocemos el mecanismo por que el que se vuelven inmunes. Ahora, un equipo del Centro Nacional de Biotecnología (CNB/CSIC) ha aplicado una molécula capaz de destruir esos mecanismos.
La célula de una bacteria es como una naranja. Se hace fuerte gracias a su piel, que sería su pared celular. Un antibiótico la pela, sus gajos se deshacen y muere. Las células humanas no tienen algo igual. Hay bacterias resistentes que fabrican paredes (o piel) con enorme efectividad. Sin embargo, necesitan para ello de unas construcciones de proteínas inmunes al antibiótico. Al dejar el motor de las bacterias sin aceite, éstas no pueden generar más piel. Así es como funcionan las moléculas que acaban con la inmunidad de las superbacterias | Vídeo: M. Viciosa y CSIC
La mayoría de antibióticos ataca a la proteína encargada de fabricar la pared celular de la bacteria. Sin embargo, se ha detectado "una proteína extra que es determinante en la fabricación y mantenimiento de esa cáscara", señala a El Independiente el doctor Daniel López, firmante del estudio publicado en Cell.
Esa proteína trabaja en un compartimento concreto y con lípidos. "Es como un motor de coche. Si destruimos el aceite de ese motor, la dejamos desprotegida". La metáfora del aceite viene muy al caso, puesto que la molécula utilizada en el CNB ataca a las llamadas balsas de lípidos (término que asociamos a las grasas). Es el lugar de la bacteria donde se ensamblan proteínas que forman los complejos que le sirven de fortaleza, una especie de zona de obras. “Estos microdominios en la membrana de la célula son clave porque en ellos se forman muchos complejos proteicos relacionados con la resistencia a los antibióticos", aclara López.
El método funciona con sustancias como las que combaten el colesterol
¿Con qué retiramos esas balsas de lípidos? Muchas de estas moléculas desarrolladas son las mismas que se prescriben, en determinados casos, para tratar el colesterol alto. El colesterol, el fin y al cabo, es un lípido.
Este descubrimiento es tríplemente importante: uno, porque hemos visto que la célula de una bacteria no es tan distinta en cuanto a complejidad a de un animal o planta. Dos, porque al ser células compartimentadas, que conocemos bien, podemos "usar esa compartimentación para matar patógenos". Y tres, porque permite rescatar viejos antibióticos que habían caído en desuso por no ser efectivos ante las resistencias creadas por las bacterias.
De hecho, en el caso experimental del equipo del CSIC, usaron meticilina, un antibiótico que ya hacía años que era incapaz de actuar ante la Staphylococcus aureus. Esta es la típica bacteria hospitalaria que tantos quebraderos de cabeza da a los equipos médicos por su resistencia. La meticilina fue capaz de curar a los ratones infectados, depués de administrárseles la molécula que ha utilizada por el equipo de López.
Daniel López en la cámara oscura del CNB
La bacteria crea estructuras de proteína (PBP2a) "que usa como andamiajes" para fortalecer su pared celular. La molécula desarrollada "rompe esos parches o balsas y así no se pueden ensamblar". Ese pretratamiento abre el camino a la acción de la penicilina, que ya no encuentra resistencia al topar con el patógeno. La bacteria deja de ser súper.
Ya no descubriremos más antibióticos

En 1960 el Celbenin (meticilina) era anunciado como un gran avance
El avance, publicado en Cell, resulta clave en un momento en que estamos casi en puertas de regresar a la era pre-Flemming. Al año se estima que las bacterias resistentes a antibióticos están causando 25.000 muertes en Europa y 700.000 en el mundo. Naciones Unidas ha llamado a la acción. Los laboratorios no sintetizan estas medicinas de la nada. Hay que buscarlas en la naturaleza. Y ya no nos queda por dónde mirar.
"Cuando hacía el postdoctorado, en Estados Unidos –rememora López– uno de mis mentores me decía que ya no había más antibióticos. Que, como mucho, se podía rastrear el el mar en busca de alguna bacteria nueva rara y su antibiótico, pero, aún así, lleva muchos años ver que no sea tóxica en humanos, que funcione... es muchísimo dinero".
Por el contrario, los antibióticos clásicos en desuso "son muy baratos".
Podría funcionar en bacterías como la legionela
El equipo trabaja ya en poder trasladar estos resultados prometedores con la Staphylococcus aureus en otros patógenos. "Por el momento tenemos sólo estudios en modelos por ordenador", aclara López, pero hay al menos cinco bacterias candidatas a responder igual de bien a la acción de su molécula. Entre ellas, la Legionella pneumophila, responsable de muertes como las ocurridas la semana pasada en Barcelona.
Usamos en ratones meticilina, abandonada hace años por ser inútil ante bacterias resistentes a los antibióticos. Los roedores se curaron.
"Lo que ahora miramos es el genoma de las bacterias, la ruta de síntesis para esos lípidos y esa proteína, lo que indicaría que tienen la capacidad de formar esos parches" y, por tanto, ser igual de efectivos.
¿Cabe la posibilidad de que la bacteria mutase de nuevo y se hiciera resistente al tratamiento con esta molécula? Podría ser, pero no es probable. Las bacterias mutan porque ven amenazada su propia vida. El método que plantea el CNB suprime las balsas de lípidos, pero no mata por sí mismo a la célula. "No afecta su supervivencia", sentencia López. En realidad, la bacteria no sabe que lo que viene después es un antibiótico. No tiene, en principio, presión biológica o necesidad de mutar.
Te puede interesar
Lo más visto
- 1 La Fiscalía pide seis años y medio de cárcel para el teniente coronel Oliva
- 2 Una actriz de Sueños de libertad anuncia su embarazo
- 3 Madrid acoge más menores inmigrantes que Cataluña
- 4 Luis García Montero y las viudas vituperadas
- 5 El presidente del Grupo Tragsa ve "asqueroso" que la exnovia de Ábalos no acudiera a su puesto de trabajo
- 6 Dos bomberos mueren y otros cuatro resultan heridos en un incendio en un parking de Alcorcón
- 7 El creador de Adolescencia, serie de Netflix, desmonta su mentira
- 8 Madrid se revuelve ante las dudas del Gobierno sobre menores inmigrantes
- 9 Zapatero: "No me siento nada orgulloso de nuestra especie"